DNA-Methylierung als Biomarker für die Frühdiagnose von Lungenkrebs ausgemacht
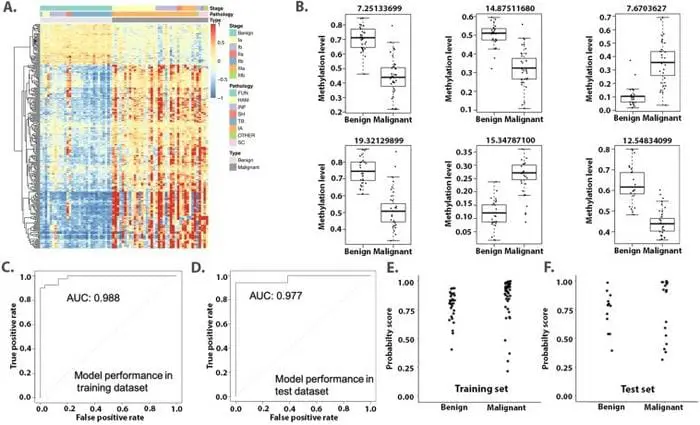
In einer aktuellen Studie, die in der Zeitschrift Genes and Diseases veröffentlicht wurde, untersuchten Forscher der Universität Sichuan und der Stanford University die DNA-Methylierungsprofile von ctDNA in Plasma- und Gewebeproben von Patienten mit Lungenkrebs oder gutartigen Erkrankungen mittels Bisulfit-Sequenzierung. Die Analyse identifizierte 276 unterschiedlich methylierte Marker, die spezifisch für Lungenkrebs sind.
Von den 276 vorhergesagten Markern wiesen sechs einen signifikant unterschiedlichen Methylierungsstatus zwischen Lungenkrebs und gutartigen Erkrankungen in der Gewebekohorte auf. Von diesen wurden zwei bei Lungenkrebs und vier bei gutartigen Erkrankungen als hypermethyliert befunden. In ähnlicher Weise wurden in der Plasmakohorte neun differentiell methylierte CpG-Stellen (DMS) identifiziert, von denen nur zwei bei Lungenkrebs hypermethyliert waren, während die restlichen sieben hypomethyliert waren. Ein diagnostisches Vorhersagemodell, das auf diesen Mustern basiert, war in der Lage, Lungenkrebs erfolgreich von gutartigen Erkrankungen in Trainings- und Validierungsgewebekohorten zu unterscheiden. Obwohl das diagnostische Vorhersagemodell zeigte, dass aus dem Plasma stammende Methylierungs-Biomarker die Krebsfrüherkennung unterstützen können, waren ihre Sensitivität und Spezifität geringer als die der aus dem Gewebe stammenden Marker. Außerdem wurde eine signifikante Korrelation zwischen den Delta-Methylierungswerten der Gewebe- und Plasmaproben festgestellt.

Eine auf Methylierungshaplotypen basierende Analyse identifizierte 1222 differentiell methylierte Regionen in Gewebeproben, die in mit der DNA-Replikation zusammenhängenden Pfaden angereichert sind. Darüber hinaus wurden signifikante Korrelationen zwischen differentiellen Methylierungsmustern und klinischen Merkmalen festgestellt, insbesondere zwischen Rauchern und Nichtrauchern sowohl im Gewebe als auch im Plasma.
Zusammenfassend lässt sich sagen, dass die ctDNA-Methylierung sowohl im Gewebe als auch im Plasma wirksam zwischen bösartigen und gutartigen Erkrankungen unterscheiden kann und ein großes Potenzial als Biomarker für die Frühdiagnose von Lungenkrebs besitzt. In Zukunft könnte die Integration von m ultimodalen Informationen aus CT-Scans, ctDNA-Mutationen und ctDNA-Methylierungsmustern die Sensitivität und Spezifität der Lungenkrebs-Früherkennung verbessern.
Original Paper:
Lesen Sie auch:
Onkologische Symposium „Vom Biomarker zur Therapie“ – MedLabPortal
Redaktion: X-Press Journalistenbüro GbR
Gender-Hinweis. Die in diesem Text verwendeten Personenbezeichnungen beziehen sich immer gleichermaßen auf weibliche, männliche und diverse Personen. Auf eine Doppel/Dreifachnennung und gegenderte Bezeichnungen wird zugunsten einer besseren Lesbarkeit verzichtet.




