Trainingsfreie Pipeline revolutioniert Proteinbinder-Design für Krebsbehandlung
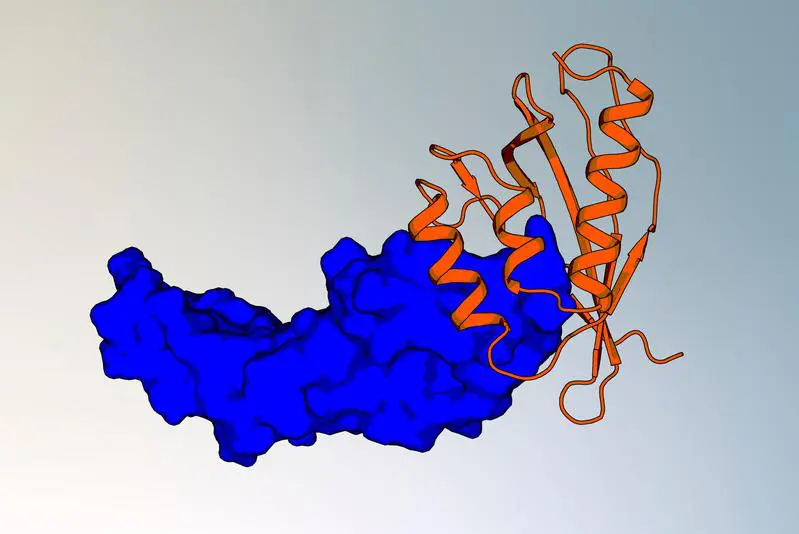
Ein Forschungsteam des Max-Planck-Instituts für Biologie Tübingen, der Universität Tübingen und des Universitätsklinikums Tübingen hat eine neuartige, trainingsfreie computergestützte Pipeline entwickelt, die präzise Proteinbinder für krankheitsrelevante Proteine entwirft.
Die in Nature Communications veröffentlichte Methode nutzt Formkomplementarität, um Binder zu erstellen, die gezielt an Zielstellen wie den Interleukin-7-Rezeptor alpha (IL-7Rα) und den Vascular Endothelial Growth Factor (VEGF) binden, die beide mit Krebs assoziiert sind. Durch Integration von Proteindatenbanken, physikbasiertem Schnittstellendesign und Molekulardynamiksimulationen wurden Binder mit hoher Bindungsaffinität, Stabilität und potenter Aktivität entwickelt.

Experimentelle Tests, einschließlich in vivo Studien, zeigten, dass die Binder Tumorwachstum in Tieren effektiv hemmen, indem sie Signalwege blockieren. Im Gegensatz zu datengetriebenen Ansätzen mit neuronalen Netzen basiert die Pipeline auf physikalischen Grundprinzipien, was ihre Anwendbarkeit auf synthetische Proteine mit künstlichen Aminosäuren erweitert.
Durch die Verwendung menschlicher Proteom-Templates werden immunogene Risiken minimiert, was die Entwicklung sicherer Therapeutika erleichtert. Die Methode vereinfacht den Designprozess, vertieft das Verständnis von Proteinfaltung und -wechselwirkungen und eröffnet neue Möglichkeiten für die Arzneimittelentwicklung sowie molekulare Diagnostik bei Krebs und Immunerkrankungen.
Original Paper:
Redaktion: X-Press Journalistenbüro GbR
Gender-Hinweis. Die in diesem Text verwendeten Personenbezeichnungen beziehen sich immer gleichermaßen auf weibliche, männliche und diverse Personen. Auf eine Doppel/Dreifachnennung und gegenderte Bezeichnungen wird zugunsten einer besseren Lesbarkeit verzichtet.




