Neue Einblicke in molekulare Maschinen durch biophysikalische Methoden
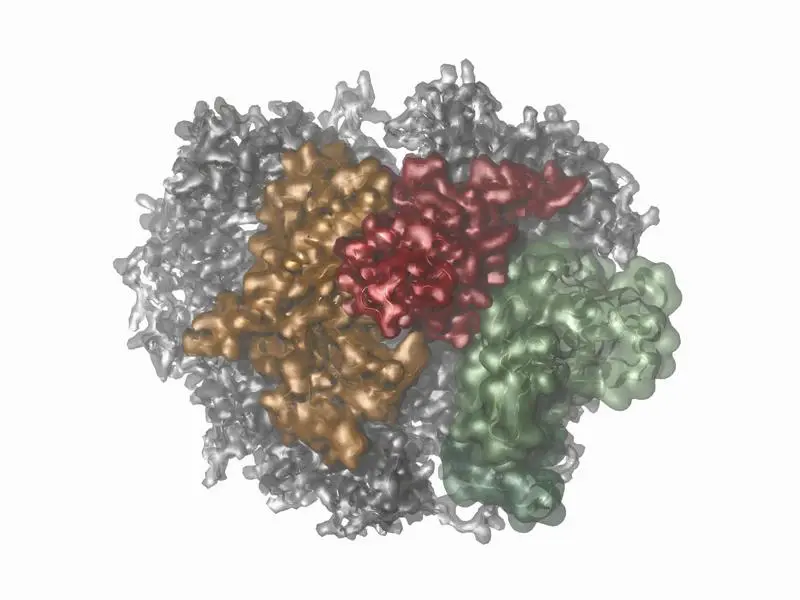
Forschende der Universität Regensburg haben bahnbrechende Einblicke in die Bewegungen des RNA-Exosom-Komplexes gewonnen, einer molekularen Maschine, die für den Abbau von RNA in Zellen verantwortlich ist. Durch die Kombination von Kryo-Elektronenmikroskopie, Kernspinresonanz-Spektroskopie (NMR) und Molekulardynamik-Simulationen (MD) gelang es dem interdisziplinären Team, die Struktur und Dynamik dieses Proteinkomplexes detailliert zu untersuchen. Die Ergebnisse, veröffentlicht in der Fachzeitschrift Nature Communications, markieren einen methodischen Fortschritt in der Erforschung großer molekularer Maschinen.
Proteine sind essenziell für sämtliche Lebensprozesse und bilden oft Komplexe, die als molekulare Maschinen vielfältige Aufgaben erfüllen, wie den Auf- und Abbau von Biomolekülen oder den Transport innerhalb von Zellen. Das RNA-Exosom, bestehend aus zehn Proteinen, spielt eine zentrale Rolle beim Abbau von RNA, einem unverzichtbaren Prozess in jeder Zelle. Während die statische Struktur solcher Komplexe durch Techniken wie die Kryo-Elektronenmikroskopie gut untersucht werden kann, war die Erfassung ihrer Bewegungen bisher eine Herausforderung. Das Team der Regensburg Center for Biochemistry (RCB) und des Regensburg Center for Ultrafast Nanoscopy (RUN) hat nun erstmals die dynamischen Prozesse des Exosoms sichtbar gemacht.

Mit Hilfe der NMR-Spektroskopie untersuchten die Forschenden, wie sich die Struktur der Proteine während ihrer Funktion verändert. Diese Methode, die normalerweise bei kleineren Proteinen eingesetzt wird, konnte durch innovative Ansätze auch auf den großen Exosom-Komplex angewendet werden. Molekulardynamik-Simulationen ergänzten die NMR-Daten, indem sie anschauliche Modelle der Proteinbewegungen lieferten. So konnten die Wissenschaftler Regionen des Komplexes identifizieren, die bisher für andere Methoden unsichtbar waren, und deren Bewegungen analysieren – von extrem schnellen Schwingungen im Milliarden-Sekunden-Bereich bis hin zu langsameren Bewegungen, die etwa 30-mal pro Sekunde auftreten.
Besonders aufschlussreich war die Entdeckung, dass einige Proteinbereiche sich in einem Rhythmus bewegen, der mit der Geschwindigkeit des RNA-Abbaus korreliert. Dies deutet darauf hin, dass die Dynamik der Proteine für ihre Funktion entscheidend ist. Die Studie liefert damit nicht nur neue Erkenntnisse über den RNA-Abbauprozess, sondern etabliert auch eine Methode, um die Bewegungen großer molekularer Maschinen zu visualisieren. Dieser Ansatz könnte die Forschung an anderen Proteinkomplexen revolutionieren und die Grundlage für ein tieferes Verständnis der dynamischen Prozesse in Zellen schaffen.
Original Paper:
4D structural biology–quantitative dynamics in the eukaryotic RNA exosome complex | Nature Communications
Redaktion: X-Press Journalistenbüro GbR
Gender-Hinweis. Die in diesem Text verwendeten Personenbezeichnungen beziehen sich immer gleichermaßen auf weibliche, männliche und diverse Personen. Auf eine Doppel/Dreifachnennung und gegenderte Bezeichnungen wird zugunsten einer besseren Lesbarkeit verzichtet.




