Bahnbrechende Methode beschleunigt bakterielle Genomevolution
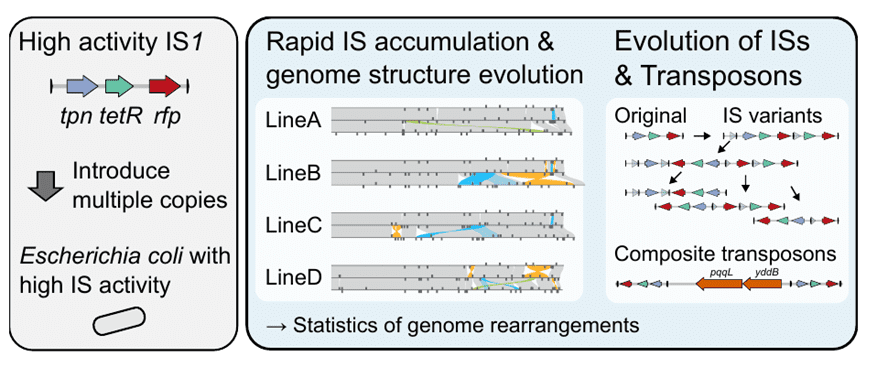
Ein Forschungsteam der Universität Tokio hat ein innovatives System entwickelt, das die Evolution der Genomstruktur in Bakterien gezielt beschleunigt. Die Methode, die sich auf kleine, mobile DNA-Sequenzen – sogenannte Insertionssequenzen (IS) – konzentriert, ermöglicht es, groß angelegte Veränderungen in der Anordnung von Genen direkt im Labor zu beobachten. Die Ergebnisse könnten neue Erkenntnisse über evolutionäre Prozesse liefern und Anwendungen in Biotechnologie und Materialwissenschaften eröffnen.
Die Struktur von Genomen, also die Anordnung der Gene in der DNA, ist entscheidend für die Funktionen von Organismen. Insertionssequenzen, auch als „springende Gene“ bekannt, verändern ihre Position im Genom und treiben so evolutionäre Veränderungen voran, etwa durch Mutationen oder Veränderungen der Genomgröße. In Escherichia coli (E. coli), einem wichtigen Modellorganismus, sind solche IS-Transpositionen jedoch selten und treten etwa einmal jährlich auf, was ihre Untersuchung erschwert.

Das Team um Yuki Kanai entwickelte eine Methode, um diesen Prozess drastisch zu beschleunigen, indem mehrere Kopien hochaktiver ISs in E. coli eingeführt wurden. Inspiriert wurde die Forschung durch Studien zu Insekten-Bakterien mit stark verkleinerten Genomen, die viele Transposons enthalten. Innerhalb von nur zehn Wochen zeigten die Testorganismen signifikante Veränderungen: etwa 25 neue IS-Einfügungen sowie eine Vergrößerung oder Verkleinerung des Genoms um über fünf Prozent – eine Geschwindigkeit, die in der Natur Jahrzehnte erfordern würde.
Die Experimente enthüllten ein komplexes Zusammenspiel von häufigen kleinen Deletionen und seltenen großen Duplikationen, das die bisherige Annahme eines einfachen Deletionsbias in der Genomverkleinerung erweitert. Zudem entstanden strukturelle Varianten und zusammengesetzte Transposons, die mögliche Evolutionspfade dieser mobilen Elemente aufzeigen. Die Ergebnisse bieten eine wertvolle Grundlage, um die Auswirkungen von IS-Insertionen, Genomgrößenänderungen und Umlagerungen auf die Fitness von Organismen zu untersuchen.
Die Studie wirft zudem neues Licht auf die Evolution der Transposons selbst, deren Rolle in der Gestaltung bakterieller Genome bislang wenig erforscht wurde. Die Methode überbrückt die Lücke zwischen der Analyse vergangener Evolution und kleinräumigen Laborveränderungen, indem sie groß angelegte Genomveränderungen in Echtzeit beobachtbar macht. Sie könnte helfen, fundamentale Fragen zu klären, etwa wie Kooperation zwischen Bakterien oder zwischen Bakterien und ihren Wirten entsteht.
Langfristig streben die Forschenden an, die Prinzipien biologischer Komplexität zu entschlüsseln. Die Methode könnte die Entwicklung komplexer Organismen oder organischer Materialien ermöglichen, die durch evolutionäre Optimierung an spezifische Funktionen angepasst werden. Die Ergebnisse eröffnen neue Perspektiven für die Grundlagenforschung und biotechnologische Innovationen.
Original Paper:
Journal article: Yuki Kanai, Atsushi Shibai, Naomi Yokoi, Saburo Tsuru, Chikara Furusawa, “Laboratory evolution of the bacterial genome structure through insertion sequence activation”, Nucleic Acids Research, https://doi.org/10.1093/nar/gkaf331
Lesen Sie auch:
Genom-Mining: Neue Wege zur Herstellung bioaktiver Wirkstoffe – MedLabPortal
Redaktion: X-Press Journalistenbüro GbR
Gender-Hinweis. Die in diesem Text verwendeten Personenbezeichnungen beziehen sich immer gleichermaßen auf weibliche, männliche und diverse Personen. Auf eine Doppel/Dreifachnennung und gegenderte Bezeichnungen wird zugunsten einer besseren Lesbarkeit verzichtet.




