Infektionsforschung setzt auf Mini-Lungen und neue Sensoren
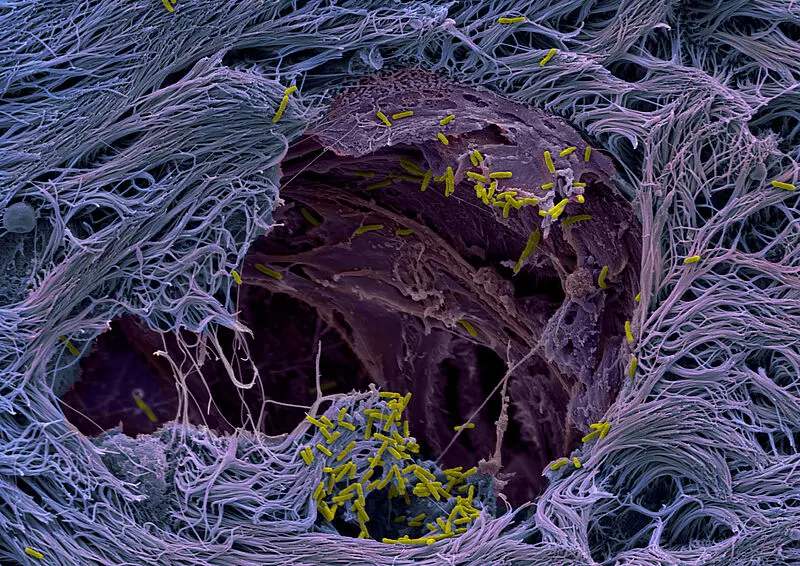
Die Erreger haben vielfältige Strategien entwickelt, um die Lunge und den Körper zu infizieren. Forschende um Urs Jenal vom Biozentrum der Universität Basel haben nun mithilfe von Mini-Lungen, welche sie im Labor aus menschlichen Stammzellen entwickelt haben, neue Erkenntnisse über das Infektionsgeschehen gewonnen. In der Fachzeitschrift Nature Microbiology beschreiben sie, wie es Pseudomonas gelingt, die oberste Schicht des Lungengewebes zu durchbrechen und in tieferliegende Bereiche vorzudringen. Die Arbeit entstand im Rahmen des Nationalen Forschungsschwerpunktes NCCR „AntiResist“.
„Wir haben Mini-Lungen gezüchtet, die den Infektionsverlauf in Patientinnen und Patienten ziemlich realitätsnah abbilden“, erklärt Jenal. „So sind wir dem Erreger auf die Spur gekommen. Er nutzt die Becherzellen, die den Schleim produzieren, als Eintrittspforte, als Trojanische Pferde. Obwohl diese Becherzellen nur einen kleinen Teil der Lungenschleimhaut ausmachen, gelangen die Keime durch sie hinter die Abwehrlinie und öffnen die Tore.“
Mit einem ganzen Waffenarsenal, sogenannten Sekretionssystemen, dringen die Keime gezielt in die Becherzellen ein, vermehren sich dort und töten die Zellen schliesslich. Weil die Zellen platzen, entsteht eine Lücke im Zellverband – die Barriere wird undicht. Diese Schwachstelle nutzen die Erreger sofort aus: Sie rücken in die Lücke vor und breiten sich in tiefere Gewebeschichten aus. Dort sind Bakterien nur schwer für Abwehrzellen oder Antibiotika zu erreichen.
Neuer Sensor zum Beobachten von Bakterien
Mithilfe der Mini-Lungen gelang es den Forschenden die raffinierte Taktik von Pseudomonas aufzuklären. Unklar ist allerdings, wie die Erreger ihr Verhalten im Verlauf der Infektion anpassen. Sie müssen beispielsweise erst mobil sein, sich bei Kontakt mit Zellen dann schnell festheften können und später ihr Waffenarsenal hochfahren. Man weiss, dass die Keime ihr Verhalten dank kleiner Signalmoleküle rasch ändern können. Bislang fehlten jedoch technische Hilfsmittel, um die genauen Zusammenhänge zu untersuchen.
Jenals Team hat einen Sensor entwickelt, mit dem sie ein bestimmtes Signalmolekül namens c-di-GMP in einzelnen Bakterien messen und verfolgen können. Die Methode ist im Fachjournal «Nature Communications» beschrieben.
„Dies ist ein technologischer Durchbruch“, so Jenal. „Jetzt können wir in Echtzeit und mit höchster Auflösung sehen, wie dieses Signalmolekül im Verlauf der Infektion reguliert wird und wie es die Virulenz des Erregers steuert. Wir können jetzt also beobachten, wann einzelne Erreger bestimmte Programme anschalten, sich zum Beispiel erst ans Gewebe festsetzen und es später angreifen. Mit der Methode können wir Lungeninfektionen noch genauer auf den Grund gehen.“
Mit Organmodellen nah an Patientinnen und Patienten
„Dank der Mini-Lungen verstehen wir nun viel besser, wie sich die Krankheitserreger im Gewebe und vermutlich auch in Patientinnen und Patienten verhalten“, sagt Jenal. Mit diesen Lungenmodellen lässt sich laut Jenal auch die Wirkung von Antibiotika im Gewebe erforschen, etwa wo die Bakterien während der Behandlung überleben. Solche Organmodelle werden in Zukunft unverzichtbar sein, wenn es darum geht, neue und effektive Strategien zur Bekämpfung von Krankheitserregern zu entwickeln.
Original Paper:
Lesen Sie dazu auch:




